日常瞎掰
对于ChIP-seq、ATAC-seq等这样捕获基因组富集区域(即分析结果中peak)的技术,大家多多少少应该有所耳闻。在分析这类测序数据的时候,必不可少的步骤就是将peak注释到基因组上,以便了解peak出现在哪些基因的周边区域,从而发现生物学上的意义。目前,注释peak的软件不在少数,如ChIPseeker、homer2等。今天我们主要来说说如何利用ChIPseeker绘制peak的分布饼图和条形图。
注释
ChIPseeker的用法相当简单,这里就顺便简单介绍一下,下面使用包里面的数据演示一下:
- 使用R里面的数据库
library(ChIPseeker)
library(TxDb.Hsapiens.UCSC.hg19.knownGene)
library(org.Hs.eg.db)
files 如上面所示,调用一下annotatePeak函数注释就完成了。其中,TxDb.Hsapiens.UCSC.hg19.knownGene数据库是必须的,org.Hs.eg.db数据库为可选项,如果提供了注释结果里面会多出ENSEMBL、SYMBOL、GENENAME三列信息。当然,annotatePeak还有其他参数可以根据需要设置,这里就不展示了。
- 使用gtf文件
library(GenomicFeatures)
txdb 这种方式注释起来也非常方便。不论使用哪种注释方法,最终生成的注释结果都是以csAnno类对象的方式存储,后续操作就可以基于这个对象了。
过滤
注释后,我们可以根据需求对其进行过滤,筛选想关注的区域来看看分布情况,比如,这里只保留Promoter、Exon、Intron三个大类的结果,可以看出过滤后每个Feature的比例会重新计算:
annofilter 当然,在过滤之前,我们要先知道都有哪些列可以用来过滤,可以使用下面的命令来查看:
head(peakanno@anno)
GRanges object with 6 ranges and 11 metadata columns:
seqnames ranges strand | V4 V5 annotation
|
[1] chr1 815093-817883 * | MACS_peak_1 295.76 Promoter (2-3kb)
[2] chr1 1243288-1244338 * | MACS_peak_2 63.19 Promoter (
[1] 1 803451 812182 8732 2 284593
[2] 1 1243994 1247057 3064 1 126789
[3] 1 2976181 2980350 4170 2 440556
[4] 1 3547331 3566671 19341 2 49856
[5] 1 3816968 3832011 15044 1 100133612
[6] 1 6304252 6305638 1387 1 390992
transcriptId distanceToTSS
[1] uc001abt.4 -2911
[2] uc001aed.3 0
[3] uc001aka.3 0
[4] uc001ako.3 0
[5] uc001alg.3 0
[6] uc009vly.2 613
-------
seqinfo: 24 sequences from hg19 genome
可视化
- barplot
使用ChIPseeker注释后,我们可以使用其内置的函数plotAnnoBar来展示样本的peak分布情况,适合一个或多个样本。这里我们选取两个样本来演示:
alist 结果如下:
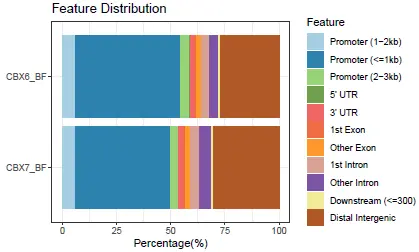
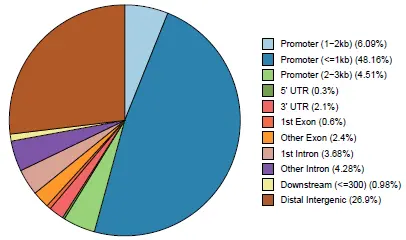
- pieplot
单个样本的时候也可以选择饼图来展示结果,调用函数plotAnnoPie即可绘图:
plotAnnoPie(peakanno)
结果如下:
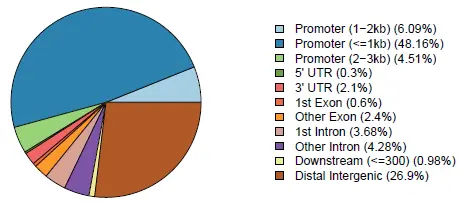
可视化函数借用
其实,我们也可以只用ChIPseeker的可视化函数来展示自己的结果而不用其来注释。如果我们已经有了注释的结果,可以将统计结果整理成类似上面所示展示的Genomic Annotation Summary数据框格式,然后借助ChIPseeker包的绘图函数来绘图:
- barplot
df1 结果如下:
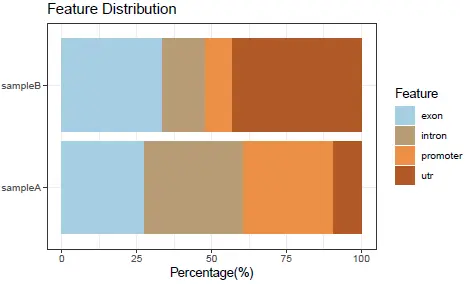
如果觉得这个barplot风格不错,想用于绘制自己的数据,借用一下函数是不是很方面快捷。准备数据的时候只需注意两点:一是必须有Feature、Frequency两列,列名不能错,分别用于指定类别及比例;二是Feature的数据类型为factor。第三列做为样本列,名称随意,如果没有categoryColumn设置为1。
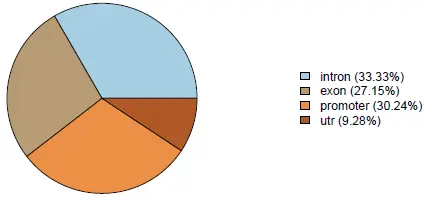
- pieplot
准备饼图数据时,只需要有Feature、Frequency两列既可,有没有其他列不会影响绘图:
df1
Feature Frequency sample
1 intron 33.333333 sampleA
2 exon 27.147766 sampleA
3 promoter 30.240550 sampleA
4 utr 9.278351 sampleA
col 结果如下:
- 自定义pieplot
其实,ChIPseeker绘制的饼图个人不是很喜欢,有两个小的因素。首先,其绘制饼图时不是基于ggplot2,而是基于基础函数pie,扩展性没有那么方便;再者,绘出的图和图例的间距比较大。虽然这两点可以忽略不计,但是如果能绘出更接近成品图的效果那后续不是更省时省力么。所以,个人借用ChIPseeker的配色方法包装了一个基于ggplot2的绘制饼图的函数,可以接受ChIPseeker的注释对象或者含有Feature、Frequency两列的数据框。下面一起来看看绘图效果:
plotpie 结果如下:
结束语
ChIPseeker做为Y叔的作品,使用体验依然是顺滑流畅没话说,功能也是非常全面强大。网上有很多关于ChIPseeker的帖子,这里列举一个本人觉得写得比较详细的帖子可供参考:https://www.jianshu.com/p/c76e83e6fa57。工欲善其事必先利其器,好的工具可以让我们事半功倍,节省更多的时间去关注数据本身的意义。不过,没事的时候学习一下工具的原理及实现过程还是有好处的,这样用起来才会更加得心应手来去自如。好了,今天到此结束~~~
往期回顾
R语言书籍免费领
可视化:网络图
可视化:Wordcloud
可视化:Dumbbell Chart
可视化:Arc Diagrams
转载请注明:OBA博客 » ChIPseeker绘图函数借用